我想使用 KDE 来估计我在显微镜图像中检测到的 XY 点列表中的簇密度(这是一个完全不同的过程)。我正在尝试调整这个答案中的代码:https://stackoverflow.com/a/64499779/2009558
为什么 KDE 的输出不映射到输入维度?我不明白将 KDE 输出映射到网格的需要是什么。也不知道为什么网格的尺寸与输入数据不匹配。这一行中“128j”的值是多少?
gx, gy = np.mgrid[x.min():x.max():128j, y.min():y.max():128j]
那是什么样的Python对象?它既有数字又有字母,但不是字符串?我尝试用谷歌搜索这个但找不到答案。Numpy 有时是如此不符合 Python 风格,这让我发疯。
到目前为止,这就是我所处的位置。数据只是一个 pandas df,其中 X 和 Y 坐标为浮点数。
import numpy as np
import plotly.express as px
import plotly.offline as offline
import pandas as pd
from scipy.stats import gaussian_kde
xx = df['X']
yy = df['Y']
xy = np.vstack((xx, yy))
kde = gaussian_kde(xy)
gx, gy = np.mgrid[xx.min():xx.max():128j, yy.min():yy.max():128j]
gxy = np.dstack((gx, gy))
# print(gxy[0])
z = np.apply_along_axis(kde, 2, gxy)
z = z.reshape(128, 128)
fig = px.imshow(z)
fig.add_trace(go.Scatter(x = xx, y = yy, mode='markers', marker = dict(color='green', size=1)))
fig.show()
这会生成我想要的大部分图:上面覆盖有点的密度图,但密度数据的尺寸为 128 x 128,而不是输入限制的尺寸。
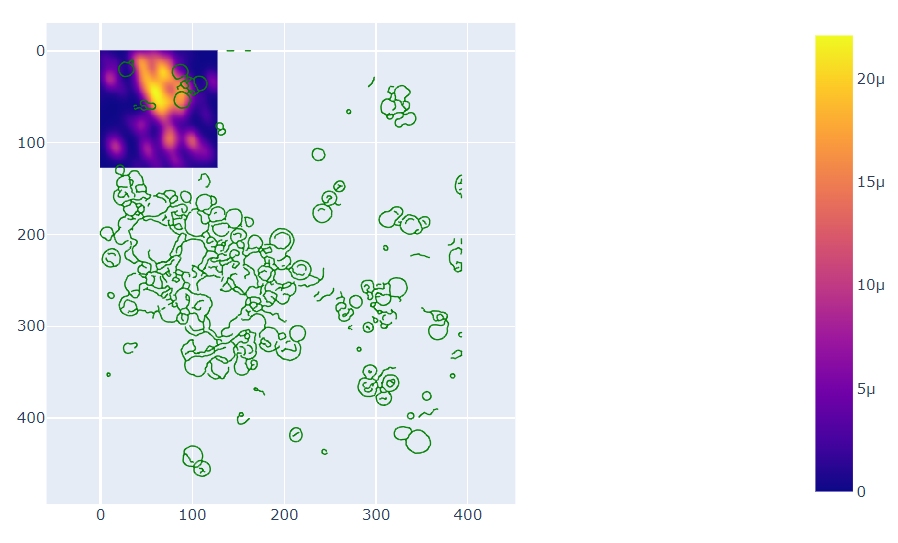
当我尝试像这样替换重塑中的真实尺寸时
z = z.reshape(ceil(xx.max()-xx.min()), ceil(yy.max()-yy.min()))
我只是收到错误。
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
~\AppData\Local\Temp/ipykernel_19840/2556669395.py in <module>
12 z = np.apply_along_axis(kde, 2, gxy)
13 # z = z.reshape(128, 128)
---> 14 z = z.reshape(ceil(xx.max()-xx.min()), ceil(yy.max()-yy.min()))
15
16 fig = px.imshow(z)
ValueError: cannot reshape array of size 16384 into shape (393,464)
先回答你的问题:
这种需求来自于将结果绘制为图像的愿望,从而绘制为像素阵列。网格在两个维度上使用常规步骤映射数据空间(尽管每个维度都有不同的步骤),然后您可以使用它来计算每个像素的颜色。
确实如此,但唯一
px.imshow(z)知道的是矩阵z,两个轴都指的是矩阵坐标,这在这里令人困惑。这是一个任意选择,将定义图像的分辨率。这里是 128 x 128,因为你的数据空间是这样划分的:
gx, gy = np.mgrid[xx.min():xx.max():128j, yy.min():yy.max():128j]但你可以选择其他任何东西。
正如slotthrop和Daraan在评论中指出的那样,这只是一个 numpy 约定,用于精确确定您是否希望包含间隔的端点。
关于您的错误,它来自于您试图将向量
z(由于之前的空间划分方式而具有 128 x 128 个元素)重新组织为 393 x 464 元素的矩阵。建议的解决方案:
您的问题源于对和
px.imshow(z)没有任何线索。为了解决这个问题,我们将使用支持xarray的事实,这将使我们将矩阵中的数据链接到它们的坐标。xyplotlyz(x, y)然后你在通话中使用
da而不是这样!zimshow